¿GPX Móvil o Pulsera? Un paseo, dos registros, random forest y R
En este post encontrarás las herramientas principales de R para el análisis de archivos .gpx (trackpoints, waypoints, routes...) y su aplicación en la comparación de registros entre pulseras de actividad con GPX y el registro móvil con Guru Maps. Compararemos los registros con los caminos oficiales de OpenStreetMaps. Además, en la segunda parte del artículo, exploraremos algunas herramientas de análisis de imagen y detección automática de elementos a través de imágenes mediante el análisis de ortofoto por color y también a través de modelos entrenados. Contenido:- Introducción
- Parte 1: Análisis de datos
- Parte 2: Experimento de teledetección. Digitalización Automática del Camino con Ortofoto
- Parte 3: Síntesis para vagos (sin acritud), pero con otro ejemplo
- Conclusiones
¿Qué método de registro gps es mejor, un móvil o una pulsera de
actividad? Con esta motivación parte este artículo. Se ha la misma ruta
pero con dos dispositivos distintos y después se ha utilizado R para
analizar esos archivo .gpx. Después, ya con el pique, se han comparado
los registros de los tracks con los caminos disponibles en
OpenStreetMaps. Por último, se ha probado a digitalizar de forma
semiautomática los caminos a través de una ortofoto, logrando algunos
resultados interesante con un esfuerzo mínimo. El resultado te
sorprenderá.
Los tracks y ortofotos utilizados en este ejercicio, por si los quieres salsear tú mismo, te los dejo aquí.
Introducción
Siguiendo el camino de conectar cosas, ¿cómo podría hacer una review tecnológica, disfrutar de la naturaleza y analizar datos geográficos con R? En plena era de la información, la clave está en la sistematización e integración. Así que, en este artículo, voy a intentar comparar los datos GPS registrados durante una ruta (y una segunda ruta extra) utilizando dos dispositivos: un Xiaomi Note 10 Pro (mi móvil de trabajo) y un reloj Amazfit Bip U Pro.
El objetivo real no es la review, ni el análisis, si no mostrar como
cualquier tema puede ser analizado y conectado con nuestros intereses.
Si además alguien ha tenido alguna vez la curiosidad de saber qué
dispositivo gps es mejor para registrar una ruta, bienvenido sea. Y digo
más, si alguien ha tenido alguna vez interés en analizar los tracks
.gpx con las librerías de R, aquí encontrarás las funciones
principales para hacerlo.
Algunas especificaciones previas:
- Xiaomi Mi Note 10
Pro:
- GPS: A-GPS, GLONASS, GALILEO, BDS - Peso: 193 gramos
- Reloj Amazfit BIp U
Pro:
- GPS: GPS + GLONASS - Peso: 24,7 gramos
Programas de registro utilizados:
-
Guru Maps Pro: para mí sigue siendo la mejor aplicación de registro de rutas y mapas offline. Tiene además los fondos de landscape y hiking son realmente buenos para hacer rutas de senderismo.
-
Zepp + Amazfit: la ruta, aunque ha sido registrada con la propia pulsera, después hay que exportarla a
.gpxdesde la aplicación Zepp.
Las rutas a comparar
La ruta principal que ha realizado para este análisis será una circular partiendo de la campa de Ucieda en pleno Parque Natural del Saja Besaya, subiendo todo al rededor del monte entre los ríos Bayones y el Regato de la Toba.
La zona de partida, por si a alguien le interesa algo más que los tracks gps y las chorradas de R:
Además, una forma de entender el contexto en el que estamos es a partir de una fotografía aérea 360 tomada bastante centrada a la ruta:
Parte 1: Análisis de datos
Preparación de los Datos
Primero, cargaremos los datos GPX registrados por ambos dispositivos y los recortaremos a la zona coincidente para evitar discrepancias debidas a diferencias en el inicio o fin de la grabación.
Lo primero, como siempre es establecer los parámetros. Incluimos en este apartado también las librerías de análisis que vamos a usar durante el proceso.
library(sp); library(ggplot2); library(osmdata); library(mapview)
library(leaflet); library(dplyr); library(opendataes); library(raster)
library(stars); library(gridExtra); library(ClusterR);library(tictoc)
library(terra); library(knitr); library(kableExtra); library(sf)
library(htmlwidgets); library(patchwork)
# gpxdir
tifdir <- "./_post_R/data/20250115/tif/"
gpxdir <- "./_post_R/data/20250115/gpx/"
Para ver qué capas componen un conjunto vectorial, en este caso un
archivo .gpx lo mejor es usar directamente st_layers(). La función
tabla no existe, simplemente es una función oculta de este documento
que permite maquetar de forma más elegante los datos de esta y otras
tablas que vayan a ir apareciendo.
# Ver capas que componene el gpx
f <- paste0(gpxdir, "20241013_01_xiaomi.gpx")
tabla(st_layers(f) %>% dplyr::select(-crs))
| name | geomtype | driver | features | fields |
|---|---|---|---|---|
| waypoints | Point | GPX | 0 | 23 |
| routes | Line String | GPX | 0 | 12 |
| tracks | Multi Line String | GPX | 1 | 13 |
| route_points | Point | GPX | 0 | 25 |
| track_points | Point | GPX | 15313 | 27 |
Para este análisis necesitaremos tanto los tracks como los
track_points. Al fin y al cabo son estos los que realmente contienen
toda la inforamción de la ruta. Usaremos el siguiente sistema de
codificación: x para xiaomi (archivo saliente de guru maps) y a
para amazfit. Después el número de archivo (contador 00). Después la
codificación t para el track y p para los trackpoints.
Verás también que utilizo los %>% desde el principio. Esto permite
reducir mucho la longitud del código en horizontal y permite leerlo
mejor y saber cual es la cadena lógica de herramientas que están
operando. Es la forma habitual que imita la secuencia de procesos que
seguirías en un model builder o algo así. En esta primera secuencia
verás que (1) se compone la ruta del archivo, (2) se lee la ruta del
archivo y (3) se crea un objeto sf. Así con cada ruta. Lo mismo para
los tracks y los trackpoints.
# archivos 1 xiaomi-gurumaps
x01t <- paste0(gpxdir, "20241013_01_xiaomi.gpx") %>%
st_read(layer = "tracks", quiet = TRUE) %>%
st_as_sf()
x01p <- paste0(gpxdir, "20241013_01_xiaomi.gpx") %>%
st_read(layer = "track_points", quiet = TRUE) %>%
st_as_sf()
# archivos 1 amazfit
a01t <- paste0(gpxdir, "20241013_01_amazfit.gpx") %>%
st_read(layer = "tracks", quiet = TRUE) %>%
st_as_sf()
a01p <- paste0(gpxdir, "20241013_01_amazfit.gpx") %>%
st_read(layer = "track_points", quiet = TRUE) %>%
st_as_sf()
Veamos alguno de ellos para ver el nivel de corrección. No te lo vas a creer, pero al registro del Redmi le he puesto el color “red” (rojo), y al registro de la pulsera, el azul, porque sí:
En este primer vistazo, sale excesivamente bien parada la ruta grabada con la pulsera, pero, respecto al móvil, el problema radica en que el gps creo que ha sido “liquidado” en alguna caida. Veo demasiadas imprecisiones desde hace tiempo y creo que se ha debido estropear algo internamente que afecta a la localización.
Ahora que ya tenemos los archivos cargados vamos a buscar el intervalo de tiempo común y filtrar las rutas para mostrar solo el intervalo de tiempo común. Como somos humanos y podemos encender y apagar el registro en tiempos distintos, para poder hacer una comparativa lo más justa posible, debemos restringir los registros a las partes comunes. Por ejemplo, para el tiempo de inicio, lo que se hace es seleccionar el valor más alto de tiempo registrado entre el inicio del registro del móvil y de la pulsera. Este sería el tiempo común de inicio. La lógica equivalente para el final. Si no se entiende prueba a hacerte un dibujito :-), os escríbeme si quieres más detalles.
# Encontrar el intervalo de tiempo común
inicio_comun <- max(min(x01p$time), min(a01p$time))
fin_comun <- min(max(x01p$time), max(a01p$time))
# Filtrar rutas al intervalo de tiempo común
x01p_cut <- x01p[x01p$time >= inicio_comun & x01p$time <= fin_comun, ]
a01p_cut <- a01p[a01p$time >= inicio_comun & a01p$time <= fin_comun, ]
Análisis de la Densidad de Puntos
Un aspecto del análisis de datos inicial podría ser el cálculo de la densidad de puntos registrados por cada dispositivo en la zona coincidente y comparar cuál tiene mayor frecuencia de registro. Vamos a calcular aquí el número de puntos (trackpoints) tomados por minuto y dispositivo.
# Calcular duración de la ruta en segundos
duracion <- as.numeric(difftime(fin_comun, inicio_comun, units = "secs"))
# Calcular número de puntos
n_puntos_xiaomi <- nrow(x01p_cut)
n_puntos_amazfit <- nrow(a01p_cut)
# Calcular densidad de puntos (puntos por minuto)
densidad_xiaomi <- n_puntos_xiaomi / (duracion / 60)
densidad_amazfit <- n_puntos_amazfit / (duracion / 60)
# Mostrar densidad de puntos
cat("Densidad de puntos Xiaomi:", densidad_xiaomi, "puntos/minuto\n")
## Densidad de puntos Xiaomi: 71.86853 puntos/minuto
cat("Densidad de puntos Amazfit:", densidad_amazfit, "puntos/minuto\n")
## Densidad de puntos Amazfit: 43.80783 puntos/minuto
Análisis del nivel de precisión
Observamos que el móvil tiene una densidad de captura de casi el doble, pero seguimos teniendo el problema de la imprecisión. Si observamos todos los datos asociados al registro de trackpoints en el gpx podemos ver que hay diferencias entre los dispositivos:
Tabla de registro gpx Guru Maps (móvil)
tabla(head(x01p_cut, 3), height = 200)
| track_fid | track_seg_id | track_seg_point_id | ele | time | magvar | geoidheight | name | cmt | desc | src | link1_href | link1_text | link1_type | link2_href | link2_text | link2_type | sym | type | fix | sat | hdop | vdop | pdop | ageofdgpsdata | dgpsid | gom_speed | geometry | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 139 | 0 | 0 | 138 | 317.937 | 2024-10-13 11:24:28 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 3 | 6 | NA | NA | NA | 1.116 | POINT (-4.219009 43.23563) |
| 140 | 0 | 0 | 139 | 318.074 | 2024-10-13 11:24:29 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 3 | 6 | NA | NA | NA | 1.168 | POINT (-4.218994 43.23563) |
| 141 | 0 | 0 | 140 | 317.868 | 2024-10-13 11:24:31 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 3 | 6 | NA | NA | NA | 1.201 | POINT (-4.218977 43.23562) |
Tabla de registro gpx pulsera Amazfit
tabla(head(a01p_cut, 3), height = 200)
| track_fid | track_seg_id | track_seg_point_id | ele | time | magvar | geoidheight | name | cmt | desc | src | link1_href | link1_text | link1_type | link2_href | link2_text | link2_type | sym | type | fix | sat | hdop | vdop | pdop | ageofdgpsdata | dgpsid | geometry |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 0 | NA | 2024-10-13 11:24:28 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | POINT (-4.219122 43.23553) |
| 0 | 0 | 1 | NA | 2024-10-13 11:24:29 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | POINT (-4.219099 43.23555) |
| 0 | 0 | 2 | NA | 2024-10-13 11:24:30 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | POINT (-4.219069 43.23557) |
Como podemos observar, el móvil registra otros campos de interés, como la ele, la gom_speed, la hdop y la vdop. ¿y qué son estos? Ni idea, pero si le pregunto a ChatGPT nos lo dirá:
📍 1. ele (Elevation)
- Significado: Altitud o elevación del punto respecto al nivel del mar (en metros).
- Uso: Se utiliza para calcular desniveles, perfiles altimétricos y datos relacionados con la altura.
📍 2. gom_speed
- Significado: Velocidad del dispositivo en movimiento en un momento específico (en km/h o m/s).
- Nota: Este campo no es estándar en los archivos GPX y parece una variable personalizada por el software o dispositivo que generó el archivo. Podría ser la velocidad instantánea registrada en cada punto.
📍 3. hdop (Horizontal Dilution of Precision)
- Significado: Precisión horizontal del GPS, medida en metros.
- Detalles:
- Un valor bajo indica buena precisión.
- Un valor alto indica que el GPS tiene una mala precisión debido a la geometría de los satélites.
- Rango típico:
- <1: Excelente
- 1-2: Muy buena
- 2-5: Aceptable
- >5: Pobre
📍 4. vdop (Vertical Dilution of Precision)
- Significado: Precisión vertical del GPS, medida en metros.
- Detalles:
- Al igual que el hdop, un valor bajo indica buena precisión.
- La precisión vertical suele ser peor que la horizontal debido a la distribución de los satélites en el cielo.
No contaba con hacer este análisis, pero una vez que sabemos que al menos el móvil sí recoge estos datos de precisión, ¿podemos analizar la precisión de la captura a partir de la precisión de estos datos?
Analicemos la distribución de valores de precisión en hdop y vdop:
p1 <- ggplot(x01p_cut, aes(x = hdop)) +
geom_histogram(binwidth = 0.5, fill = "#55A5D3", color = "white") +
theme_minimal() +
labs(x = "HDOP", y = "Frecuencia") +
xlim(0, 10)
p2 <- ggplot(x01p_cut, aes(x = vdop)) +
geom_histogram(binwidth = 0.5, fill = "#55A5D3", color = "white") +
theme_minimal() +
labs(x = "VDOP", y = "Frecuencia") +
xlim(0, 10)
p1 / p2
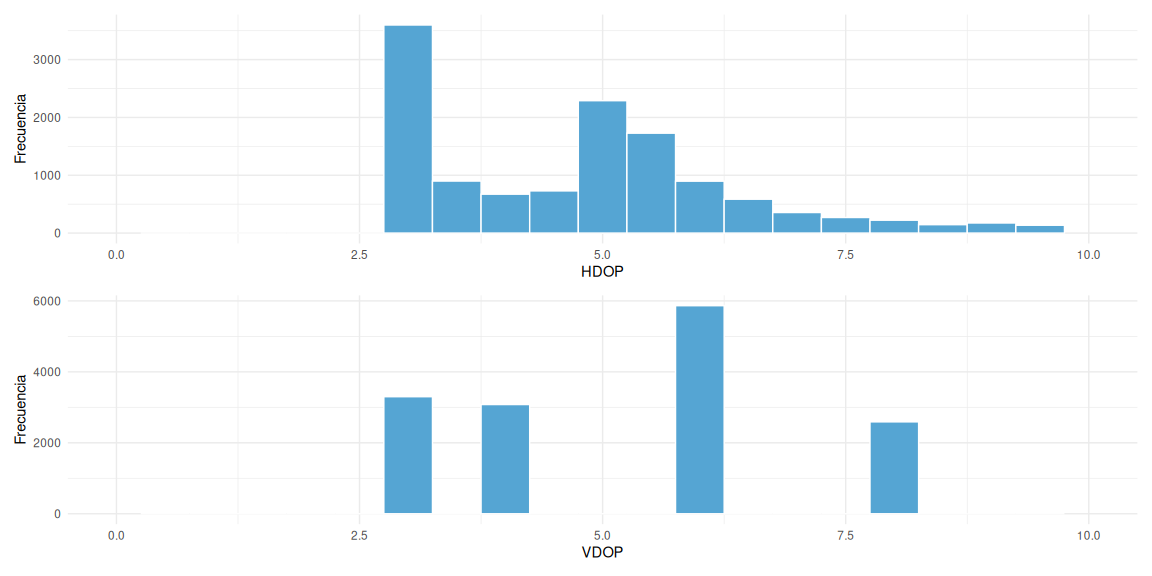
Estos parámetros no lo había estudiando nunca antes pero podrían ser interesantes para mucha gente. Si hacemos caso a los rangos de niveles de precisión, observamos efectivamente, que la extraña sinuosidad de las líneas trazadas por el móvil pueden deberse efectivamente a los valores relativamente altos de hdop y vdop, situándose todos los valores entre los rangos “aceptable” y “pobre”. Estaría bien analizar estos datos en un Garmin, ¿verdad? Eso lo dejaremos para cuando vuelva a tener uno.
Comparación con Datos de OpenStreetMap (OSM)
¿Cómo podemos saber si estos datos se ajustan a la realidad?
Utilizaremos datos de sendas y carreteras de OSM para determinar
cuál de los dos dispositivos se ajusta más a la verdad. Ojo, es posible
que los trazados de los senderos descargables de OSM hayan sido
“fabricados” a partir de las rutas gpx grabadas por vete tú a saber qué
dispositivos y digitalizadas y corregidas después sobre ortofoto.
Inevitablemente, las cosas se hacen así, y no hay topógrafos ni dinero
suficiente para digitalizarlo todo de forma real y precisa.
Sin embargo, el mundo de la captura satelital sumada a la inteligencia artificial ha hecho que cada vez se pueda digitalizar de forma más precisa y a menor coste (tranquilos, los topógrafos seguirán siendo necesarios). Aquí os dejo algunos ejemplos:
- Extracción Automática de Redes Viales desde Imágenes Satelitales
de Alta Resolución
PDF:
- Estudio: Brightearth Roads: Towards Fully Automatic Road Network Extraction from Satellite Imagery (2024).
- Resumen: Este trabajo propone una metodología completamente automatizada para extraer redes viales a partir de imágenes satelitales de muy alta resolución. Utiliza una red neuronal convolucional para la segmentación de caminos, seguida de algoritmos de optimización para convertir las predicciones en vectores lineales, y un modelo de aprendizaje automático para clasificar los materiales de las vías.
- Predicción de Redes Viales con Datos Multimodales Impulsada por
IA PDF:
- Estudio: AI Powered Road Network Prediction with Multi-Modal Data (2023).
- Resumen: Este estudio introduce un enfoque innovador para la detección automática de caminos mediante aprendizaje profundo, fusionando imágenes satelitales de baja resolución con datos de trayectorias GPS. Se investigan estrategias de fusión temprana y tardía, evaluando el rendimiento en diferentes arquitecturas de modelos y dominios geográficos.
- Extracción de Geometría de Límites de Carriles desde Imágenes
Satelitales
PDF:
- Estudio: Lane Boundary Geometry Extraction from Satellite Imagery (2020).
- Resumen: Este trabajo presenta un método para modelar mapas de alta definición de autopistas utilizando segmentación píxel a píxel en imágenes satelitales, complementando enfoques basados en nubes de puntos LIDAR y ofreciendo una alternativa más económica y rápida.
Tras este paréntesis informativo, vamos a lo que vamos.
La siguiente propuesta es, primero, delimitar el bounding box de los
datos de OSM que vamos a descargar, segundo descargar los datos en forma
de líneas (viales)y tercero mostrar todo, las tres fuentes, a la vez
en un mismo mapa para entender lo realizado.
# Definir bounding box de la zona común
bbox <- st_bbox(rbind(x01p_cut %>% dplyr::select(), a01p_cut %>% dplyr::select()))
# Obtener datos de OSM
osm_datos <- opq(bbox = bbox) %>%
add_osm_feature(key = 'highway') %>%
osmdata_sf()
# Extraer carreteras
vias <- osm_datos$osm_lines
Por probar otro tipo de mapa, que también puede servir para muchos (no
paneable y más sintético y cerrado), vamos a verlo esta vez con
ggplot:
ggplot() +
geom_sf(data = vias, color = 'yellow', size = 3) +
geom_sf(data = x01t, color = 'red', size = 1, alpha = 0.5) +
geom_sf(data = a01t, color = 'blue', size = 1, alpha = 0.5) +
ggtitle("Comparación de Rutas GPS con OSM") +
theme_minimal()
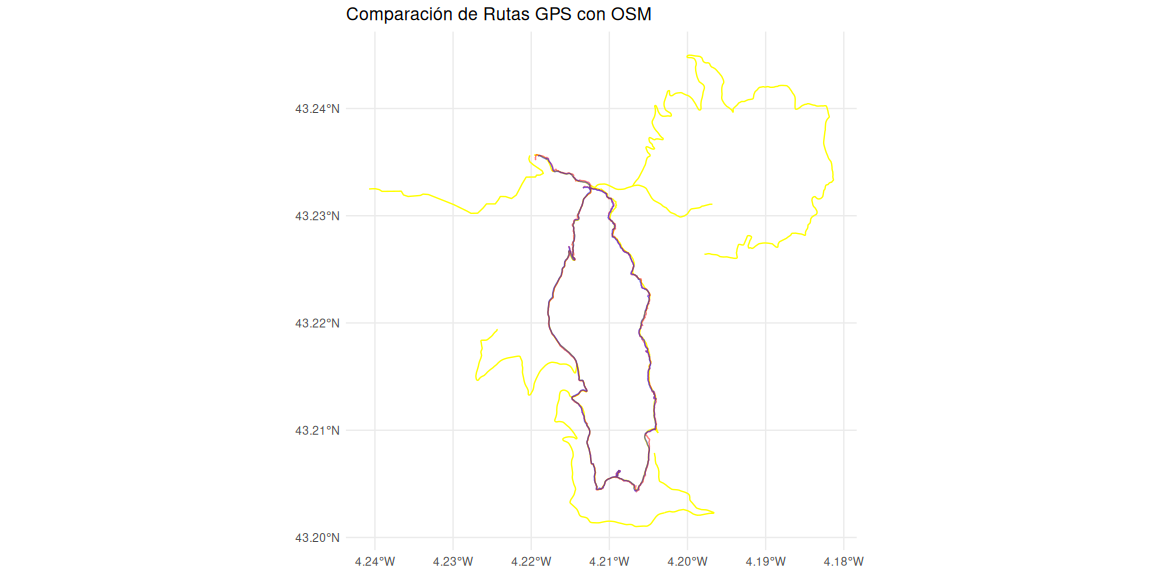
Con esto no podemos ver lo destalles, pero por lo menos son sirve como comprobación rápida de que los tracks grabados existen en OSM en todo su recorrido. Vamos a ver ahora, sobre ortofoto, como se comportan los tres tracks a la vez:
Si vas siguiendo las líneas, verás que en las partes donde hay las carreteras cláramente definidas y visibles a través del dosel tienen una digitalización casi perfecta en OSM, sin embargo, en las zonas de monte a tarvés o campo donde no está claramente el curso del paso de las personas (y animales), pues OSM tiene algunos fallos (sobre todo en la parte este de la ruta).
En el siguiente apartado vamos a asumir que OSM es dios, la realidad absoluta, la verdad, la verificación, y vamos a realizar un análisis de discrepancia geográfica, concepto que me acabo de inventar.
Análisis de Ajuste a la Realidad
Mediremos la distancia de cada punto registrado por los dispositivos a las carreteras de OSM para cuantificar el ajuste a la realidad.
# Calcular distancia mínima de cada punto a las carreteras
dist_xiaomi <- st_distance(x01p, vias)
dist_amazfit <- st_distance(a01p, vias)
# Obtener la distancia mínima para cada punto
min_dist_xiaomi <- apply(dist_xiaomi, 1, min)
min_dist_amazfit <- apply(dist_amazfit, 1, min)
# Promedio de distancias mínimas
mean_dist_xiaomi <- mean(min_dist_xiaomi)
mean_dist_amazfit <- mean(min_dist_amazfit)
cat("Distancia media Xiaomi a carreteras OSM:", mean_dist_xiaomi, "metros\n")
## Distancia media Xiaomi a carreteras OSM: 6.497789 metros
cat("Distancia media Amazfit a carreteras OSM:", mean_dist_amazfit, "metros\n")
## Distancia media Amazfit a carreteras OSM: 5.533312 metros
Con este dato tampoco es que hagamos mucho, pero parece al menos que la ruta registra por la pulsera Amazfit tiene menos distancia a la registrada en OSM.
Vamos a visualizar estos datos ahora en forma de boxplots para ver también la discrepancias entre los datos. Además, para fliparnos un poco, vamos hacer un test de hipótesis para certificar de forma cutre si las diferencias entre los registros son significativas.
Para facilitar las cosas y como los conjuntos tienen longitudes distintas se va a realizar una pequeña corrección sobre los datos: - Seleccionar de los conjuntos el percentil 95 de los datos. - Seleccionar al azar los valores dentro de ese nuevo conjuto para homogeneizar el número de datos den los conjuntos.
# Calcular los percentiles 2.5 y 97.5 para ambos conjuntos
q025_xiaomi <- quantile(min_dist_xiaomi, 0.025)
q975_xiaomi <- quantile(min_dist_xiaomi, 0.975)
q025_amazfit <- quantile(min_dist_amazfit, 0.025)
q975_amazfit <- quantile(min_dist_amazfit, 0.975)
# Filtrar los valores dentro del 95% central
filtered_xiaomi <- min_dist_xiaomi[min_dist_xiaomi > q025_xiaomi & min_dist_xiaomi < q975_xiaomi]
filtered_amazfit <- min_dist_amazfit[min_dist_amazfit > q025_amazfit & min_dist_amazfit < q975_amazfit]
# Homogeneizar el número de datos tomando muestras aleatorias
set.seed(123) # Fijar semilla para reproducibilidad
n <- min(length(filtered_xiaomi), length(filtered_amazfit))
sampled_xiaomi <- sample(filtered_xiaomi, n)
sampled_amazfit <- sample(filtered_amazfit, n)
# Crear un data frame para el boxplot
df_dist <- data.frame(
Dispositivo = rep(c("Xiaomi", "Amazfit"), each = n),
Distancia = c(sampled_xiaomi, sampled_amazfit)
)
# Crear el boxplot
ggplot(df_dist, aes(x = Dispositivo, y = Distancia, fill = Dispositivo)) +
geom_boxplot(outlier.color = "black", outlier.size = 0.8, outlier.alpha = 0.2,
color = "gray40", alpha = 0.2) +
scale_fill_manual(values = c("blue", "red")) +
theme_minimal() +
labs(x = "Dispositivo", y = "Distancia mínima (m)") +
theme(legend.position = "none")
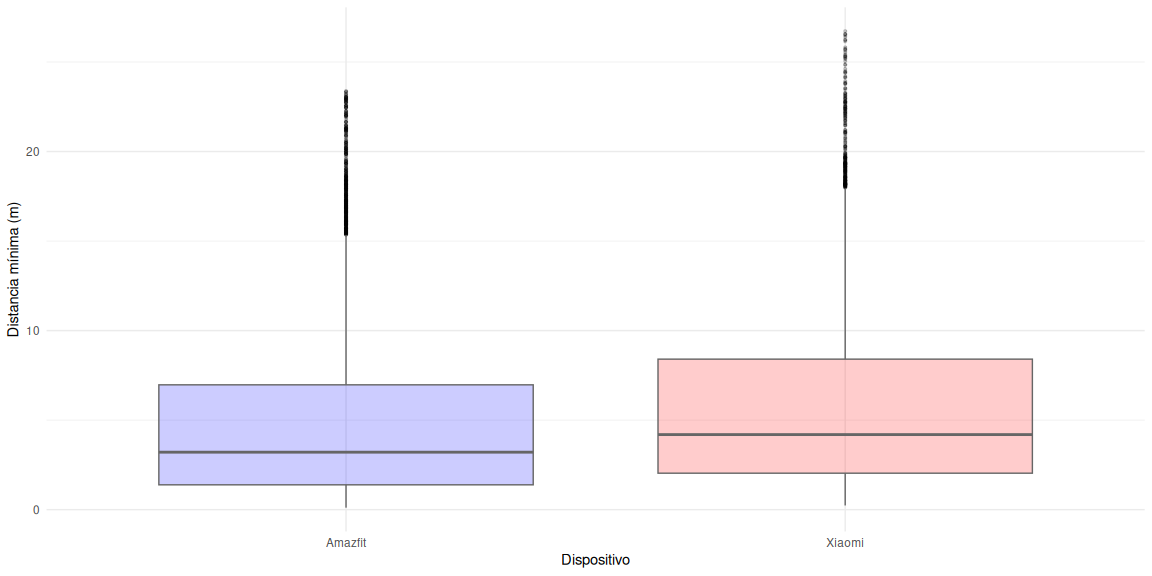
Y ahora la flipada: el test de las medias.
# Realizar el t-test de Welch sobre los datos filtrados y homogeneizados
t_test_result <- t.test(sampled_xiaomi, sampled_amazfit)
# Mostrar el resultado del t-test
print(t_test_result)
##
## Welch Two Sample t-test
##
## data: sampled_xiaomi and sampled_amazfit
## t = 11.802, df = 17582, p-value < 2.2e-16
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## 0.7194297 1.0060048
## sample estimates:
## mean of x mean of y
## 5.773363 4.910646
Esto puede servir también como recordatorio para todos aquellos que alguna vez estudiamos estadística y se nos atragantó. Interpretemos y repasemos:
El resultado indica que el test ha comparado las medias de los dos conjuntos de datos (sampled_xiaomi y sampled_amazfit) y ha encontrado diferencias estadísticamente significativas entre ellas.
-
El valor del estadístico t (12.8) indica una diferencia considerable entre las medias y los grados de libertad del test (df = 17.6) reflejan el tamaño efectivo de la muestra.
-
El p-value es extremadamente pequeño (p-value < 2.2e-16), lo que significa que la probabilidad de que esta diferencia sea producto del azar es casi nula. Rechazamos la hipótesis nula. Hay diferencias significativas entre las medias.
-
95% confidence interval: [0.805, 1.094]: El intervalo de confianza del 95% indica que la diferencia promedio entre las dos medias se encuentra entre 0.805 y 1.094 unidades. Media de sampled_xiaomi: 5.86 Media de sampled_amazfit: 4.91
La media del conjunto Xiaomi es mayor que la media del conjunto Amazfit por aproximadamente 0.95 unidades en promedio.
- Conclusión: Existe una diferencia significativa en las distancias mínimas registradas por los dispositivos Xiaomi y Amazfit. El dispositivo Xiaomi parece reportar valores más altos de distancia mínima que Amazfit. Esta diferencia no es aleatoria y se puede considerar estadísticamente significativa con un 95% de confianza.
[…]
Espera un momento, hemos usado el test de Welch pero este debería usarse si asumimos que las distribuciones de los datos son normales…y no lo son a la luz evidentemente del boxplot anterior. Por lo tanto, vamos a usar otro test que se ajustaría mejor a este tipo de conjuntos de datos. Se trata del test Wilcoxon | Mann-Whitney U test:
# Realizar el Mann-Whitney U Test (Wilcoxon Rank-Sum Test)
mw_test_result <- wilcox.test(sampled_xiaomi, sampled_amazfit, exact = FALSE)
# Mostrar el resultado del test
print(mw_test_result)
##
## Wilcoxon rank sum test with continuity correction
##
## data: sampled_xiaomi and sampled_amazfit
## W = 44043640, p-value < 2.2e-16
## alternative hypothesis: true location shift is not equal to 0
Sin más, volvemos a ver que el p-value es muy pequeño (p-value < 2.2e-16) por lo que podemos rechazar la hipótesis nula o afirmar que hay diferencias significativas en la distribución de las distancias mínimas registradas por ambos dispositivos.
GANADOR OFICIAL (MENOR DISCREPANCIA RESPECTO AL DIOS OSM): PULSERA AMAZFIT
Parte 2: Experimento de teledetección. Digitalización Automática del Camino con Ortofoto
Hemos asumido en el análisis anterior que OSM es la hostia. Sin embargo, ni las hostias son perfectas. ¿Puede una ortofoto actual ser mejor que lo digitalizado en OSM? ¿Puede además serlo teniendo en cuenta las complicadas condiciones del lugar, donde gran parte de los tramos están absolutamente tapados por el dosel?
Para empezar en este análisis en R, utilizaremos técnicas de procesamiento de imágenes para intentar digitalizar automáticamente el camino seguido. Descargaremos una ortofoto de un visor oficial y después veremos las herramientas a utilizar para la teledetección automatizada.
Nota del futuro: este análisis de la búsqueda exacta de un sendero a partir de una fotografía aérea es totalmente innecesario en caso de contar con vuelo lidar. Estos datos, de tenerlos, nos porporcionarían resolución y herramientas suficientes para peinar cada piedra debajo del dosel. Si además lo sumáramos a la clasificación automática que ya es la leche para estos datos, el análisis de imágenes resultaría estupido y totalmente innecesario. No obstante, vamos a hacerlo, porque precisamente estamos aquí para explorar caminos.
Descarga de ortofotos
Vamos a descargar y recortar ortofotos de la recientemente renovada página de CNIG. Que conste que he estado probando librerías para la descarga directa de estos datos directamente en R pero no he sido capaz. Cualquier persona que sea capaz de saltarse este paso y realizar todo el proceso de descarga directamente en R que me avise por favor.
Para descargar ortofotos de CNIG, solo tenéis que hacer esto:
Una vez descargado y para facilitar nuestro trabajo realizaremos un
recorte del tif para reducirlo a la zona de trabajo y simplificar las
cargas.
# Carga del raster
orto <- rast(paste0(tifdir, "PNOA_MA_OF_ETRS89_HU30_h25_0057_4.tif"))
# Crear el bbox original en WGS84
bbox_ext <- ext(-4.219122, -4.203974, 43.204253, 43.235632)
bbox_wgs84 <- vect(bbox_ext, crs = "EPSG:4326")
# Transformar el bbox al sistema de coordenadas del raster
bbox_orto <- project(bbox_wgs84, crs(orto))
# Agregar un buffer de 250 metros
bbox_orto_buffered <- buffer(bbox_orto, width = 250)
# Recortar el raster usando el bbox con buffer
pnoamask <- crop(orto, bbox_orto_buffered)
# Exportar el raster recortado manteniendo las coordenadas proyectadas originales
writeRaster(pnoamask, "~/Descargas/PNOA_crop_buffer.tif", overwrite = TRUE)
Cargar la imagen satelital de trabajo:
orto <- terra::rast(paste0(tifdir, "PNOA_crop.tif"))
plot(orto)

Como no necesitamos la resolución de 0.15 para este ejercicio, vamos a subir la escala un pelín. Si quieres jugar a “full HD” pues no ejecutes esto.
orto <- terra::aggregate(orto, fact = 5, cores = 10)
## |---------|---------|---------|---------|=========================================
Detección de elementos a partir de funciones de color
Detectar caminos directamente a partir del color. Este técnica es la más sencilla de ejecutar y la muestro aquí por si, en vuestros análisis crees que podéis identificar patrones directamente por el color. Obviamente, en la actualidad ya hay funciones de machine learning mil veces más potentes, pero dejo esta aquí reflejada como el origen de todo. Además, sirve también para interpretar estos ráster de PNOA, que no dejan de ser ráster multibanda, donde cada banda es un color (RGB) y el resultado es la mezcla de estos. Por ejemplo, operando sobre el color podemos identificar los caminos simplemente sabiendo que su brillo es mayor que en las zonas arboladas. Burdo pero efectivo:
# Calcular índice de brillo
brillo <- (orto[[1]] + orto[[2]] + orto[[3]]) / 3
plot(brillo)
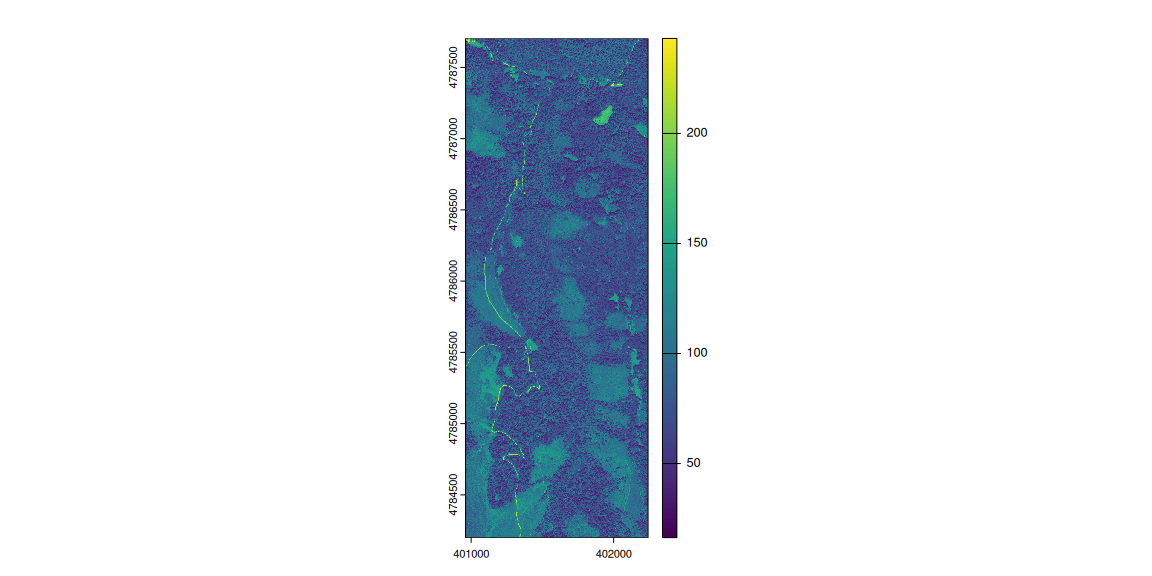
Si estuviéramos buscando identificar carreteras, seguramente, el brillo buscado sería justo al revés, y habría que buscar los tonos más oscuros. Sin embargo, aquí tenemos masa forestal con caminos de tierra, así que vamos a por brillos altos:
# Aplicar un umbral para detectar caminos
caminos <- brillo > 150 # Ajusta el umbral según tu imagen
# Plot de los caminos detectados
plot(caminos, main = "Caminos Detectados")
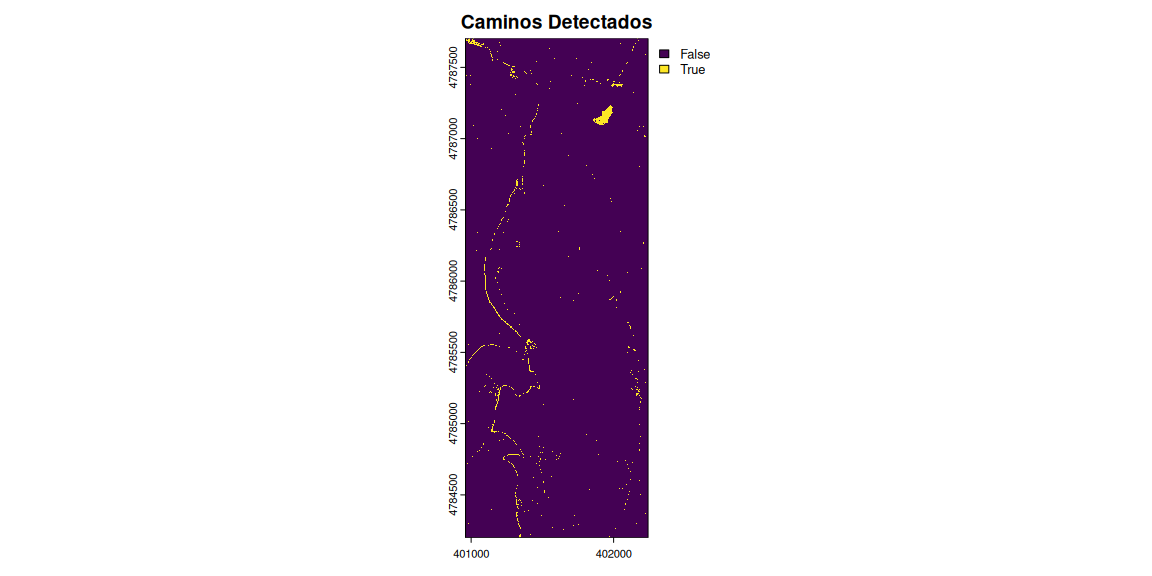
Puede parecer que no hemos hecho gran cosa, porque la herramienta es bastante rudimentaria. Ha servido más bien para detectar zonas despejadas. Puede ser útil también para detectar, simplemente a partir del color, las zonas arboladas o, si sabemos el patrón de verde de una especie concreta pues buscar directamente por el rango de verces buscados. Insisto, esta herramienta ya ha sido superada tanto por las fuentes (ver productos multiespectrales landsat) como por algoritmos automatizados de teledetección.
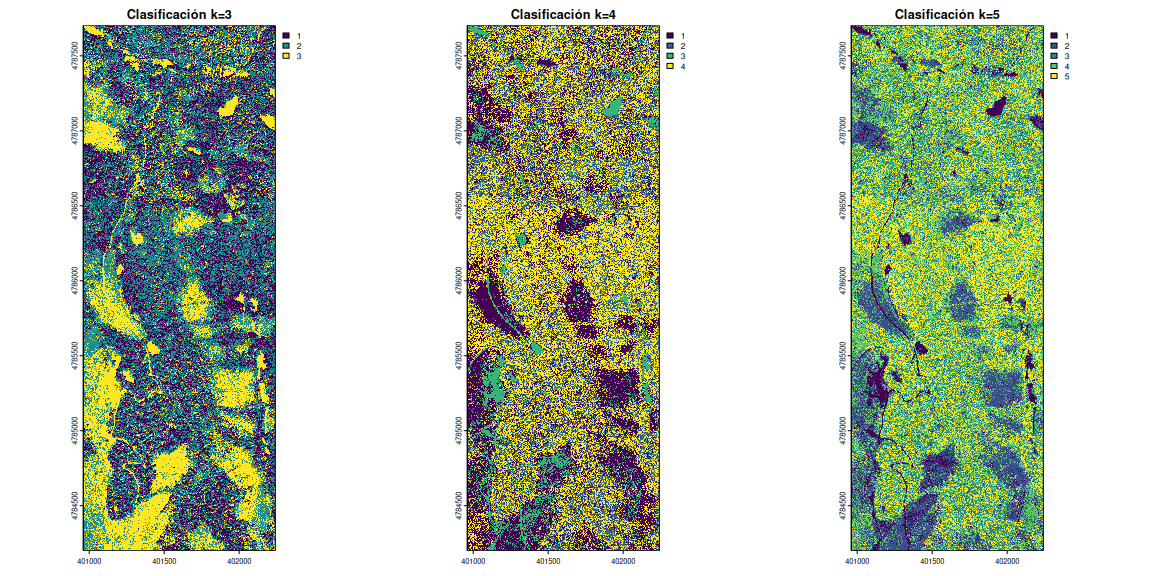
Clasificación no supervisada para noobs
Se puede realizar una clasificación de forma sencilla utilizando
herramientas de clasificación no supervisada. Es decir, no tenemos
que decidir parámetros de decisión, solo el número de grupos
diferentes que queremos detectar en la imagen. Vamos a realizar por
ejemplo la clasificación automática en 3, 4 y 5 grupos y ver si, con
esta discriminación, tenemos una categoría clara de caminos transitados.
Estos algoritmos se basan en la metodología de kmeans, pero están
hipersimplificados para hacer la búsqueda sin tener que estar
estableciendo tropecientos parámetros antes. Vamos allá.
library(RStoolbox)
library(rasterVis)
# Aplicar clasificación no supervisada directamente al raster
set.seed(123)
clasificacion_ns3 <- unsuperClass(orto, nClasses = 3)
clasificacion_ns4 <- unsuperClass(orto, nClasses = 4)
clasificacion_ns5 <- unsuperClass(orto, nClasses = 5)
# Definir el layout para mostrar tres gráficos en una fila
par(mfrow = c(1, 3))
# Realizar los gráficos
plot(clasificacion_ns3$map, main = "Clasificación k=3")
plot(clasificacion_ns4$map, main = "Clasificación k=4")
plot(clasificacion_ns5$map, main = "Clasificación k=5")
¿Y si ahora probáramos con la normalización de los datos de imagen…?
# Aplicar clasificación no supervisada directamente al raster
set.seed(123)
clasificacion_ns3 <- unsuperClass(orto, nClasses = 3, norm = TRUE)
clasificacion_ns4 <- unsuperClass(orto, nClasses = 4, norm = TRUE)
clasificacion_ns5 <- unsuperClass(orto, nClasses = 5, norm = TRUE)
# Definir el layout para mostrar tres gráficos en una fila
par(mfrow = c(1, 3))
# Realizar los gráficos
plot(clasificacion_ns3$map, main = "Clasificación k=3")
plot(clasificacion_ns4$map, main = "Clasificación k=4")
plot(clasificacion_ns5$map, main = "Clasificación k=5")
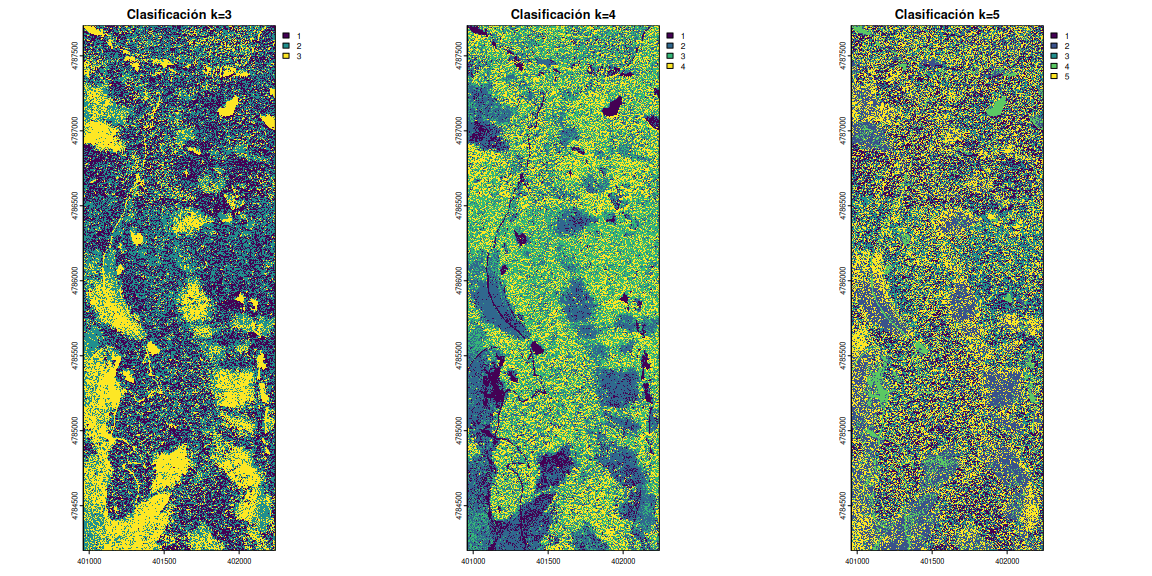
# Restaurar la configuración original de par
par(mfrow = c(1, 1))
Parece que el resultado ha mejorado bastante con la normalización. Pero
pasa una cosa, y es que *la resolución original a la que hemos
trabajado ha sido de 0.15 m, o 0.75 msi has aplicado la primera
agregación **, lo cual ha hecho que este post sea especialmente pesado.
¿Qué pasaría si directamente aplicamos una agregación a los píxeles del
ráster, lo que comunmente se llama cambiar la escala? Con la herramienta
terra::aggregate podemos cambiar la resolución del ráster. Si
seleccionamos por ejemplo usamos fact = 30 lo que estamos diciendo es
que multiplique por 30 el valor actual de resolución. Aunque hay
parámetros para cambiar el método de asignación de valores (media,
*min, max, etc) lo dejaremos por defecto. Vamos a verlo:
rsel <- clasificacion_ns4$map
rsel_reducido <- terra::aggregate(rsel, fact = 5)
## |---------|---------|---------|---------|=========================================
plot(rsel_reducido)
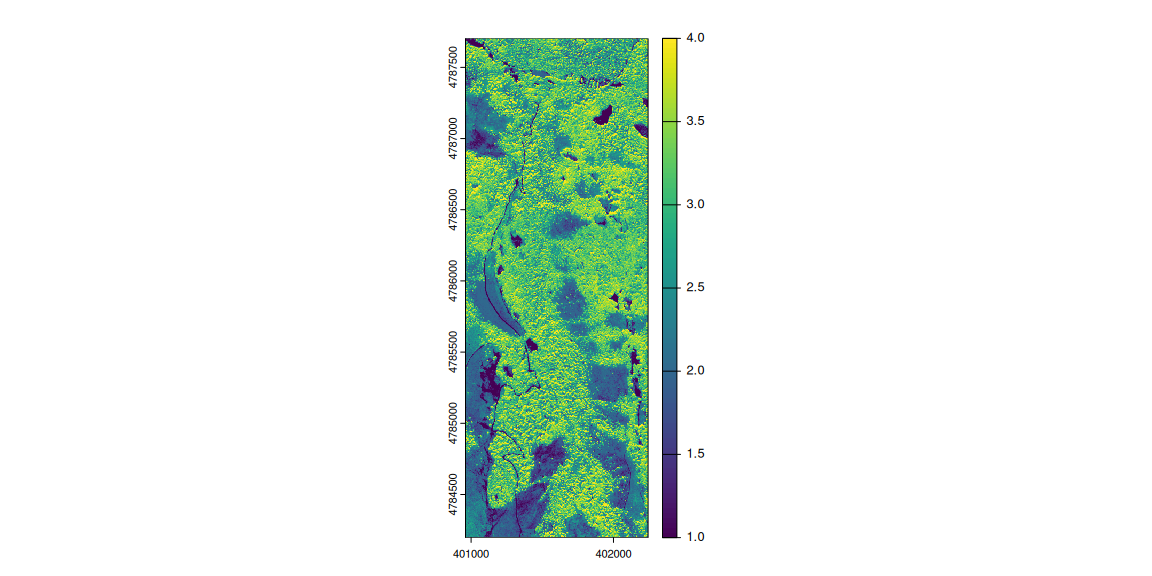
Wow. Parece que el ráster original, con todo su detalle, ahora sí se ha transformado en un ráster manejable, al menos a esta escala y con las categorías de suelo mucho más definidas. ¿Podría haberse aplicado la agregación ya desde el inicio y haber llegado al mismo resultado? Que lo haga otro. Yo me quedo con este.
Ahora sí, vemos cómo la categoría 4 se ajusta de forma fiel a los caminos de tierra y zonas despejadas. Lo bueno es que las zonas despejadas de pastos también han sido bien identificadas (clase 4) y las categorías 1 y 2 serían las zonas arboladas. No vamos a seguir profundizando en esto pero que conste que cualquiera podría ponerse a serguir buceando.
Vamos ahora a mostrar cómo luce este ráster clasificado con todo lo de antes:
Era inevitable que el sistema de clasificación solo a partir de fotografía aérea funcionara como se esperaba, sin embargo el resultado donde no hay dosel tapando es espectacular.
¿Listos para el siguiente paso?
Clasificación ráster para pros (Random Forest prediction)
Una vez entendido y aplicado el básico kmeans (que se usa para muchas
más cosas de las que puedas imaginar), es bueno saber que hay algoritmos
específicos de identificación de patrones en imagen. Puedes explorarlos
y me cuentas si alguno de ellos es capaz de resolver la parte derecha de
estre track que es donde realmente está la chicha. Algunos de ellos son:
- Filtrado por Textura (Gabor Filters)
- Análisis de Componentes Principales (PCA)
- Algoritmo de Detección de Bordes (Sobel/ Canny Edge Detection)
Pero nosotros vamos a usar uno de los también más ampliamente utilizados en multitud de temas, tanto en predicción dicotómica como en probabilidades y muchas otras cosas. Hablamos una vez más del Random Forest. Se considera en realidad un algoritmo de predicción supervisada porque tiene varios parámetros a los que hay que atender para obtener buenos resultados. Pero, ¿por qué vamos a ir a por este directamente? Pues precisamente porque tenemos la MUESTRA DE CAMINO, es decir, podemos decirle al modelo, por donde hemos registrado el camino y que se apoye en esta información para obtener los mejores resultados. ¿No sería esta la combinación perfecta entre mis piernas y una máquina?
Notas previas:
- Nota importante 1: el modelo con
randomForestpuede ser usado, como hemos comentado, tanto para predicciones de regresión como de clasificación. Ten en cuenta que tenemos que hacer una clasificación de valores, así que hay que pasarlos a factor e ir jugando con el númerontreehasta alcanzar buenos resultados. - Nota importante 2: como el conjunto de datos de la extracción de
valores a lo largo del camino es enorme por la alta resolución del
ráster, se ha introducido un muestreo aleatorio de datos para reducir
este conjunto. De no hacerlo es posible que os encontréis con este
mensaje:
Error en randomForest.default(valores_muestreados, ntree = 500, importance = TRUE): long vectors (argument 20) are not supported in .C - Nota importante 3: para no eternizar el modelo, la ortofoto se va a simplificar en escala (ver método anterior). Como ya hemos visto antes esto podría traernos buenos resultados y así aligeramos todo esto.
library(randomForest)
# orto simple
orto_simple <- aggregate(orto, fact = 6)
# Cargar la ortofoto y datos de entrenamiento (camino)
entrenamiento <- vect(a01t)
valores_camino <- terra::extract(orto_simple, entrenamiento)[,-1]
# Generar puntos aleatorios fuera del camino (no camino)
set.seed(123)
valores_no_camino <- spatSample(orto_simple, size = nrow(valores_camino), method = "random")
# Crear etiquetas para los datos
valores_camino$label <- 1 # Camino
valores_no_camino$label <- 0 # No Camino
# Combinar ambos conjuntos de datos
valores_comb <- rbind(valores_camino, valores_no_camino)
# Convertir la etiqueta en factor
valores_comb$label <- as.factor(valores_comb$label)
# Eliminar posibles NA en alguna extracción
valores_comb <- valores_comb[complete.cases(valores_comb),]
# Entrenar el modelo Random Forest
modelo <- randomForest(label ~ ., data = valores_comb, ntree = 500, importance = TRUE)
modelo
##
## Call:
## randomForest(formula = label ~ ., data = valores_comb, ntree = 500, importance = TRUE)
## Type of random forest: classification
## Number of trees: 500
## No. of variables tried at each split: 1
##
## OOB estimate of error rate: 39.3%
## Confusion matrix:
## 0 1 class.error
## 0 1752 874 0.3328256
## 1 1195 1443 0.4529947
El modelo de Random Forest entrenado es de tipo clasificación, con 500
árboles generados. La tasa de error estimada fuera de la bolsa (OOB
error rate) es del 39%, lo que indica que el modelo tiene una precisión
moderada para diferenciar entre caminos y no caminos. La matriz de
confusión muestra que el modelo identificó correctamente 2138 píxeles de
clase 0 (no camino) y 1448 píxeles de clase 1 (camino), pero también
hubo errores significativos: 1021 píxeles de clase 0 fueron clasificados
incorrectamente como clase 1, y 1721 píxeles de clase 1 fueron
clasificados como clase 0. Esto sugiere que el modelo tiene
dificultades para distinguir ciertas áreas de caminos, posiblemente
debido a similitudes en los valores de los píxeles entre ambas clases o
variabilidad en las condiciones del terreno.
¿Y si ahora comprobamos el modelo entrenado y predecimos el valor tanto en la ortofoto original como en la simplificada?
Vamos a ver primero el resultado de la predicción sobre la ortofoto simplificada:
# Predecir la clase para cada píxel del raster original
prediccion <- predict(orto_simple, modelo)
# Visualizar los caminos detectados
plot(prediccion, main = "Caminos Detectados - Random Forest")
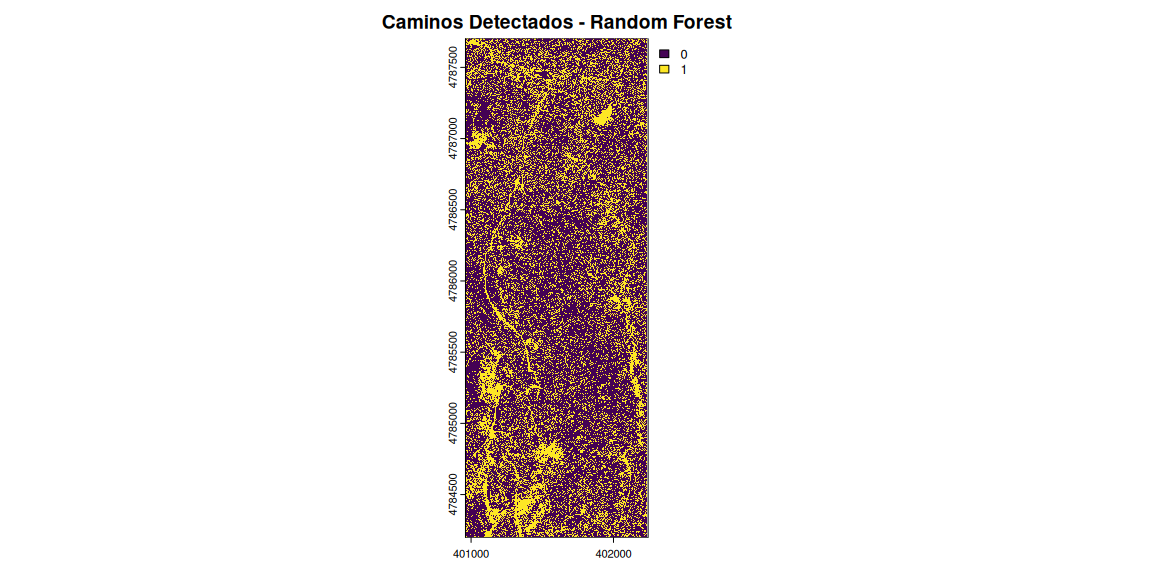
Aunque los valores del modelo han sido bastante desastrosos, sí llegamos a ver la silueta del camino en la zona este, donde por color no llegabamos a ninguna opción buena. ¿Podría ser este un buen camino? Vamos a probar a no reducir tanto la resolución de la ortofoto y ver si los resultados con mejores:
- Buffer de 0.5 m sobre la línea del camino.
- Eliminar de la muestra aleatoria los valores justo del camino.
# orto simple
orto_simple <- aggregate(orto, fact = 2)
## |---------|---------|---------|---------|=========================================
# Cargar la ortofoto y datos de entrenamiento (camino)
entrenamiento <- vect(st_buffer(a01t, 0.5))
valores_camino <- terra::extract(orto_simple, entrenamiento)[,-1]
# Generar puntos aleatorios fuera del camino (no camino)
entrenamiento2 <- project(entrenamiento, crs(orto_simple))
raster_no_camino <- terra::mask(orto_simple, entrenamiento2, inverse = TRUE)
set.seed(123)
valores_no_camino <- spatSample(raster_no_camino,
size = nrow(valores_camino),
method = "random", na.rm = TRUE)
# Crear etiquetas para los datos
valores_camino$label <- 1 # Camino
valores_no_camino$label <- 0 # No Camino
# Combinar ambos conjuntos de datos
valores_comb <- rbind(valores_camino, valores_no_camino)
# Convertir la etiqueta en factor
valores_comb$label <- as.factor(valores_comb$label)
# Eliminar posibles NA en alguna extracción
valores_comb <- valores_comb[complete.cases(valores_comb),]
# Entrenar el modelo Random Forest
modelo2 <- randomForest(label ~ .,
data = valores_comb,
ntree = 500,
importance = TRUE)
modelo2
##
## Call:
## randomForest(formula = label ~ ., data = valores_comb, ntree = 500, importance = TRUE)
## Type of random forest: classification
## Number of trees: 500
## No. of variables tried at each split: 1
##
## OOB estimate of error rate: 41.25%
## Confusion matrix:
## 0 1 class.error
## 0 31830 17788 0.3584989
## 1 23147 26471 0.4665041
# Predecir la clase para cada píxel del raster original
prediccion2 <- predict(orto_simple, modelo2)
# Visualizar los caminos detectados
plot(prediccion2, main = "Caminos Detectados - Random Forest")

Esto ha sido un absoluto desastre la verdad, pues el modelo falla más que una escopeta de feria. Pero, un segundo, muchos puntos de error están en decir que es camino lo que no lo es. Sin embargo, sí vemos claramente que el patrón del camino ha sido detectado y fuera de él lo que hay es mucho ruido. Podríamos ahora mezclar esta técnica con la de Procesamiento Morfológico (erosión y dilatación) para ver si así podríamos deshacernos del ruido y detectar patrones lineales?
Un apunte rápido de cómo va esto. La erosión reduce los grupos pequeños de píxeles al aplicar un filtro que toma el valor mínimo dentro de una ventana definida (en este caso, una matriz de 3x3). Esto elimina el ruido aislado y reduce los bordes de las formas, dejando solo las áreas más grandes y conectadas. La dilatación es el proceso inverso. Se toma el valor máximo dentro de una ventana definida, expandiendo las áreas detectadas y conectando líneas interrumpidas.
Es viejo, pero aquí podéis descargar el texto para profundizar más.
Para aplicar este concepto con la librería terra:
# Erosión: Reduce pequeños grupos de píxeles
raster_erosionado <- terra::focal(prediccion2, w = matrix(1, 3, 3), fun = min)
# Dilatación: Restaura los patrones conectando líneas
raster_dilatado <- terra::focal(raster_erosionado, w = matrix(1, 3, 3), fun = max)
A continuación podemos ver la secuencia del procesa con este último resultado final donde se aprecia la mejora sustancial en la limpieza.
# Definir el layout para mostrar tres gráficos en una fila
par(mfrow = c(1, 3))
# Realizar los gráficos
plot(terra::rast(paste0(tifdir, "PNOA_crop.tif")), main = "Ortofoto")
plot(entrenamiento2, add = TRUE, col = "red")
plot(prediccion2, main = "Predicción2")
plot(raster_dilatado, main = "Limpieza")
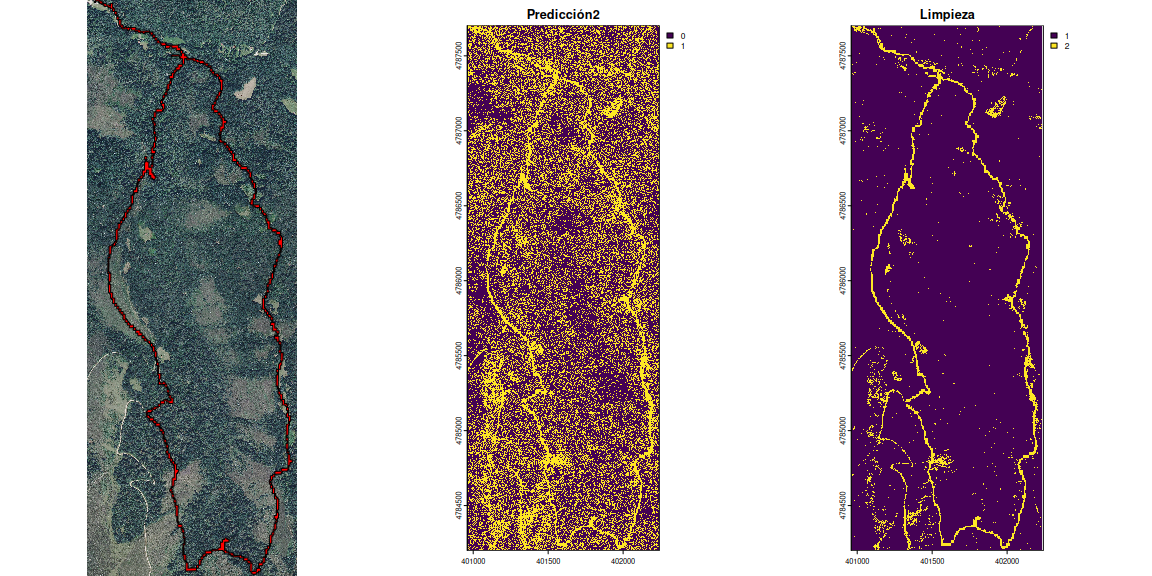
¿Cómo podemos comparar ahora esta identificación sintética del camino con el registro del gpx de la pulsera? Podríamos por ejemplo ver el % de acierto:
entrenamiento <- vect(st_buffer(a01t, 0.5))
data <- terra::extract(prediccion2, entrenamiento)[,-1]
table(data)
## data
## 0 1
## 4 49614
La tabla extraída muestra que casi todos los puntos que conforman el track de seguimiento real han sido identificados correctamente. Sin duda, se podría seguir haciendo limpieza y buscando posibilidad de generar un archivo impecable de seguimiento como de los caminos del entorno, pero es un buen avance.
Parte 3: Síntesis para vagos (sin acritud), pero con otro ejemplo
Ahora vamos a intentar centralizar en un solo proceso un análisis y
validación de una ruta realizada en monte abierto sin obstáculos ni
gargantas, con una pequeña zona arbolada en el camino de vuelta. Se
trata del ascenso al pico Liguardi en Cantabria. Podría subir más de
estas cosas si interesa, pero lo dejamos así: una fotografía aérea
montada con marzipano:
Vamos a crear funciones útiles para hacer todo esto que hemos ido desgranando pero de forma más directa:
# gpx to sf
gpxsf <- function(gpxname, layer){
paste0(gpxdir, gpxname, ".gpx") %>%
st_read(layer = layer, quiet = TRUE) %>%
st_as_sf()
}
Extraer puntos y tracks de archivos gpx:
# archivos 2 xiaomi-gurumaps
x02t <- gpxsf("20250112_Liguardi_xiaomi", "tracks")
x02p <- gpxsf("20250112_Liguardi_xiaomi", "track_points")
# archivos 2 amazfit
a02t <- gpxsf("20250112_Liguardi_amazfit", "tracks")
a02p <- gpxsf("20250112_Liguardi_amazfit", "track_points")
Crear intervalos comunes:
# Encontrar el intervalo de tiempo común
inicio_comun <- max(min(x02p$time), min(a02p$time))
fin_comun <- min(max(x02p$time), max(a02p$time))
x02p_cut <- x02p[x02p$time >= inicio_comun & x02p$time <= fin_comun, ]
a02p_cut <- a02p[a02p$time >= inicio_comun & a02p$time <= fin_comun, ]
Descargar y crear vías OSM:
# Definir bounding box de la zona común
bbox <- st_bbox(rbind(x02p_cut %>% dplyr::select(), a02p_cut %>% dplyr::select()))
osm_datos <- opq(bbox = bbox) %>% add_osm_feature(key = 'highway') %>% osmdata_sf()
viasosm <- osm_datos$osm_lines
Vistazo rápido de mapas:
# Graficar rutas y carreteras
ggplot() +
geom_sf(data = x02p_cut, color = 'red', size = 1, alpha = 0.1) +
geom_sf(data = a02p_cut, color = 'blue', size = 1, alpha = 0.1) +
geom_sf(data = viasosm, color = 'yellow', size = 3) +
ggtitle("Comparación de Rutas GPS con OSM - Ascensióna Liguardi") +
theme_minimal()
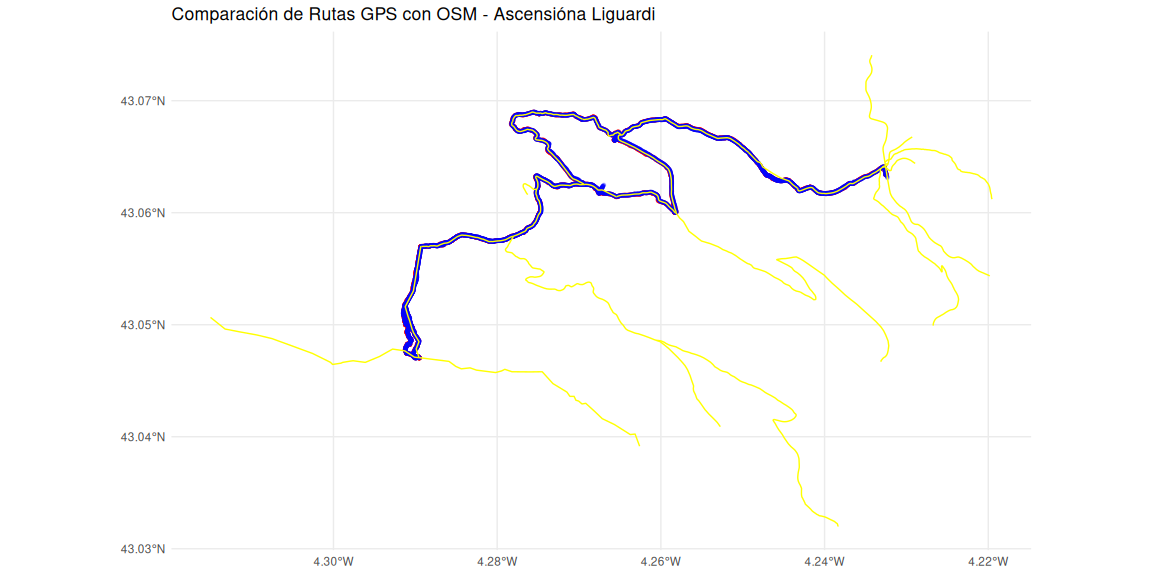
Apunte: la parte izquierda de la ascensión está fuera del track de OSM por una razón. Nos perdimos y fuimos subiendo a monte como las cabras, por encima de las piedras resbaladizas y quebradas. ¡Pa abeno matao!
De nuevo, nos flipamos (espero que por última vez) y vamos a contrastar las diferencias entre las rutas y el sendero indicado en OSM:
# función de distancia
mindisttoosm <- function(ps, vias){st_distance(ps, vias) %>% apply(1, min)}
min_dist_xiaomi <- mindisttoosm(x02p, viasosm)
min_dist_amazfit <- mindisttoosm(a02p, viasosm)
cat("Distancia media Xiaomi a carreteras OSM:", mean(min_dist_xiaomi), "metros\n")
## Distancia media Xiaomi a carreteras OSM: 9.180417 metros
cat("Distancia media Amazfit a carreteras OSM:", mean(min_dist_amazfit), "metros\n")
## Distancia media Amazfit a carreteras OSM: 6.649135 metros
Confirmamos nuevamente que el registro del móvil se desvía más del OSM peeero, apunto a un nuevo tema. Resulta que, la pulsera, cuando uno se queda quieto (en mi caso suele ser para sacar fotos, muchas fotos), deja de grabar puntos. Esto sin duda ayuda a simplificar el track y suavizarlo. Sin embargo, el móvil, está grabando todo el tiempo y, cuando uno está estático en una posición, puede haber oscilaciones no esperadas en el registro (se crean caracolillos). Esto hace que la ruta grabada sea ligeramente más larga (lo cual es irreal) y que se generen estas zonas de espiral. En la próxima ruta cambiaré las condiciones de grabación para evitar el registro en estático y que se tomen menos puntos durante la ruta, al ir caminando no es necesario ser tan constante en la grabación. No obstante, también creo que los sensores están tocados.
Antes de terminar un pequeño vistazo a la preciosidad del lugar:
Conclusiones
La comparativa de los dispositivos (sus registros gpx) no es más que
una motivación para poder analizar datos de campo de forma profunda
con R. En este post habrás visto el proceso completo desde la jornada
de campo hasta un producto. Habrás pasado por conocer la extructura de
datos de un archivo .gpx, cómo cargarlo en R, cómo trocear y limpiar
sus partes y cómo cruzar sus datos con datos de openstreetmaps.
Posteriormente hemos realizado un análisis de clasificación
supervisada y no supervisada a partir de una imagen aérea del terreno
y, la suma de los registros de campo con los de teledetección puede ser
combinada para tener digitalizaciones mucho más precisas.
Si has llegado aquí con interés en la comparativa de dispositivos en sí, hemos observado que la densidad de puntos del Xiaomi registra una mayor densidad de puntos y además tiene un atributo asociado de precisión del dato. Respecto a la pulsera Amazfit muestra una menor distancia media a las carreteras de OSM, sugiriendo una mayor precisión en la ubicación.