Lectura y procesamiento de datos .nc con R
Contenido:- Parámetros básicos
- Creación de un RASTER BASE
- Análisis de recurso eólico
- Añadir densidad poblacional
- Un experimento simple…
En este caso vamos a descargar un par de fuentes de interés y cruzar los datos, así de rápido, con el único propósito de aprender y prácticar. Vamos a manejar los datos de potencial eólico descargados en el post anterior y que pueden servirnos para hacer grandes cosas. Vamos a utilziar tres fuentes en este post:
- Ámbito de estudio. Descargaremos los límites municipales para un lugar al azar, por ejemplo…Barcelona. Fuente: CNIG.
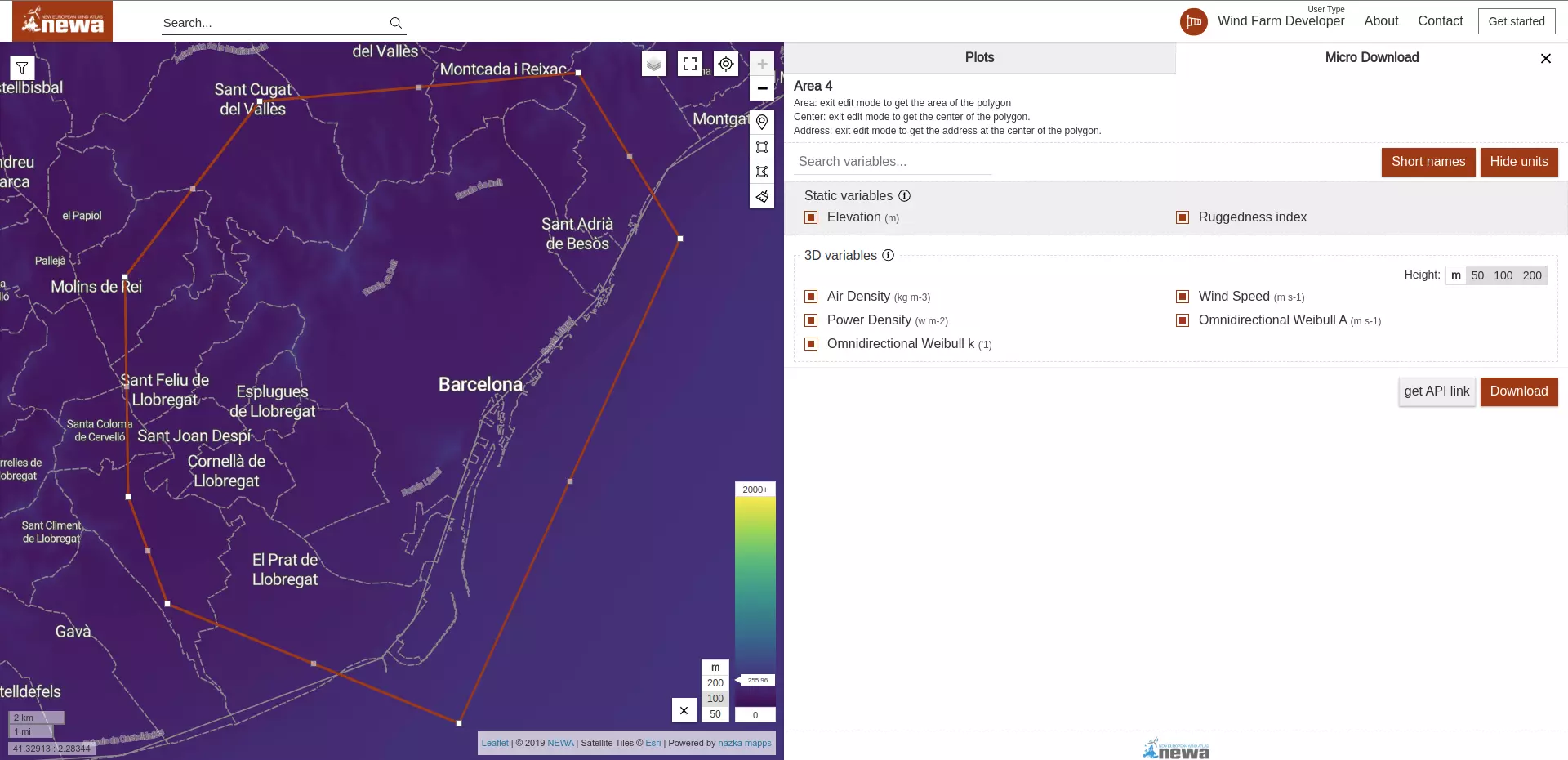
- Datos de Potencial Eólico (y todos los otros datos disponibles en la web). Fuente1: NEWA
- Datos de densidad poblacional en formato raster. Fuente: EUROSTAT.
Parámetros básicos
Como de costumbre, cargaremos las librerías que vamos a utilzar a lo
largo de este posts en un único chunk. Además, crearemos una variable
de resolución de trabajo res que servirá para el resto del documento
(interesante tenerla identificada aquí por si queremos afinar o
desafinar toda la secuencia de trabajo en el futuro).
library(raster)
library(sf)
library(rgdal)
library(dplyr)
library(fasterize)
library(terra)
library(stringr)
library(dplyr)
res <- 50 # resolución de trabajo
Veamos nuestro ámbito de estudio. En este caso, el límite administrativo del municipio de Barcelona.
amb <- read_sf("./data/20230201/AMBITO.gpkg") %>% dplyr::select()
plot(amb)
Creación de un RASTER BASE
Crear un raster de máscara que servirá como raster base para todo lo demás. Este tipo de paso es muy interesante a la hora de *homogenizar rasters**, es decir, establecer extensiones y resoluciones iguales entre capas, de forma que podamos hacer otros tipos de análisis, como los multicriterio o la preparación de datos para procesos de Machine Learning y otros.
r <- raster(extent(amb), res)
res(r) <- c(res, res)
crs(r) <- crs(amb)
amb_r <- fasterize(amb, r)
amb_r[amb_r[] == 0] <- NA
amb_r
## class : RasterLayer
## dimensions : 337, 298, 100426 (nrow, ncol, ncell)
## resolution : 50, 50 (x, y)
## extent : 922241.5, 937141.5, 4586884, 4603734 (xmin, xmax, ymin, ymax)
## crs : +proj=utm +zone=30 +ellps=GRS80 +towgs84=0,0,0,0,0,0,0 +units=m +no_defs
## source : memory
## names : layer
## values : 1, 1 (min, max)
Análisis de recurso eólico
Después de haber analizado los datos desde distintas librerías se ofrece
un sistema de lectura y tratamiento de los datos en R que facilita
muchos las cosas. Lo mejor es leer directamente los datos del archivo
.nc con la librería terra. La combinación de las librerías ncdf4 y
raster no son para nada tan eficientes y además, complican las cosas a
la hora de cargar en los rasters sus extensiones y resoluciones. Con
terra todo es mucho más fácil en este caso. Vamos allá.
Para ver las capas descargadas dentro del conjunto de datos utilizaremos la siguiente función:
eeo <-'./data/20230201/microclimate.nc'
varnames(terra::rast(eeo))
## [1] "elevation" "rix" "wind_speed" "weib_A_combined"
## [5] "weib_k_combined" "power_dens" "air_dens"
Y la siguiente para ver las unidades de cada una de las capas:
units(terra::rast(eeo))
## [1] "m" "`1`" "m s-1" "m s-1" "m s-1" "m s-1" "m s-1" "m s-1"
## [9] "" "" "" "W/m3" "W/m3" "W/m3" "kg m-3" "kg m-3"
## [17] "kg m-3"
Vamos a descargar las siguientes:
- elevation
- rix (índice de rugosidad del terreno)
- wind_speed_height=50 (velocidad media del viento a 50 m)
- wind_speed_height=100 (velocidad media del viento a 100 m)
- wind_speed_height=200 (velocidad media del viento a 200 m)
- power_dens_height=50 (densidad energética media a 50 m)
- power_dens_height=100 (densidad energética media a 100 m)
- power_dens_height=200 (densidad energética media a 200 m)
Primero convertiremos la lectura en un raster stack. Esto reduce la
cantidad de código para seleccionar individualmente cada raster. El
problema es que sustituye los = por .. No pasa nada. Este paso se
realiza para ver cómo seleccionar varias capas de interés. Normalmente
serán distintas las capas que seleccionaremos del conjunto de datos
.nc así que por esto se trabaja de la siguiente manera.
eeo <- stack(terra::rast('./data/20230201/microclimate.nc'))
eeoelev <- subset(eeo, "elevation")
eeorix <- subset(eeo, "rix")
eeows_050 <- subset(eeo, "wind_speed_height.50")
eeows_100 <- subset(eeo, "wind_speed_height.100")
eeows_200 <- subset(eeo, "wind_speed_height.200")
eeopd_050 <- subset(eeo, "power_dens_height.50")
eeopd_100 <- subset(eeo, "power_dens_height.100")
eeopd_200 <- subset(eeo, "power_dens_height.200")
Como lo más facil en el caso de los rasters homogéneos (apilables) es hacer un raster stack, eso es lo que vamos a hacer. Además, vamos a aprovechar la ocasión para simplificar el bloque de código anterior de forma que reducimos la cantidad del mismo y lo hacemos más eficiente, resultando en un limpio raster Stack con las capas de interés.
sel <- c("elevation", "rix",
"wind_speed_height.50", "wind_speed_height.100", "wind_speed_height.200",
"power_dens_height.50", "power_dens_height.100", "power_dens_height.200")
eeostack <- stack(lapply(sel, function(x) subset(eeo, x)))
eeostack
## class : RasterStack
## dimensions : 432, 427, 184464, 8 (nrow, ncol, ncell, nlayers)
## resolution : 50, 50 (x, y)
## extent : 3652800, 3674150, 2054350, 2075950 (xmin, xmax, ymin, ymax)
## crs : +proj=laea +lat_0=52 +lon_0=10 +x_0=4321000 +y_0=3210000 +ellps=GRS80 +units=m +no_defs
## names : elevation, rix, wind_speed_height.50, wind_speed_height.100, wind_speed_height.200, power_dens_height.50, power_dens_height.100, power_dens_height.200
## min values : 0.000000, 0.000000, 1.912369, 2.875424, 3.836778, 2.001225, 3.091411, 4.183888
## max values : 508.3549500, 0.1371932, 5.5574999, 5.4896860, 5.9558015, 6.0897174, 6.0551257, 6.4912443
Sin duda, este último chunk luce mucho mejor.
Ahora vamos a realizar el proceso de clip y reescalado (homogenización) de rasters, consolidando todos a partir de una capa creada anteriormente como ámbito de estudio, en la cual se establece la extensión y resolución para las celdas de análisis.
# reproyectar y "mascarar" capas
eeoelev <- mask(projectRaster(eeostack, amb_r), amb_r, updateNA = TRUE)
en caso de que se quieran guardar estas capas se podría usar la
siguiente función. Si se guardan con esta configuración, en formato
.grd, se mantendrán los nobmres de las capas y el orden de las bandas
del raster.
writeRaster(eeorasterdata, paste0(outdir, "eeodata.grd"), overwrite=TRUE)
Añadir densidad poblacional
Podríamos querer hacer un cruzamiento de datos con información raster de otro tipo. Por ejemplo, vamos a descargar una de las fuentes oficiales de densidad poblacional sobre el territorio. Por ejemplo, esta del EUROSTAT. Está actualizada en 2018 y tiene una resolución de 1x1 km². Para este ejemplo sencillo nos vale.
pd <- raster('./data/20230201/JRC_1K_POP_2018.tif')
pd <- mask(projectRaster(pd, amb_r), amb_r, updateNA = TRUE)
# agregar este último raster y representar todas las variables
rb <- stack(eeoelev, pd)
plot(rb)
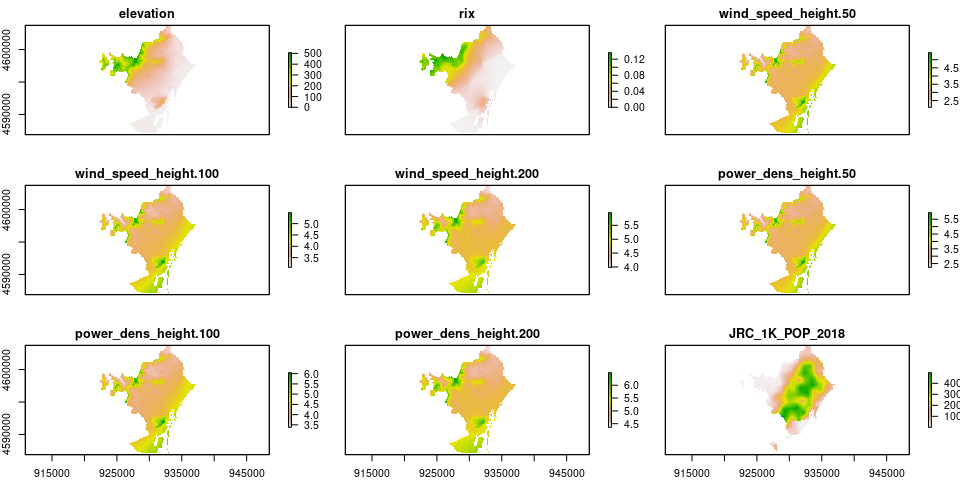
Si buscas en el conjunto de variables de este ejercicio empezarás a encontrar relaciones lógicas entre la distribución de las personas y las características naturales del medio.
Un experimento simple…
Vamos a reducir el conjunto de datos y hacer un data.frame con todos
los valores de todos los rasters seleccionados. De esta forma se
obtienen una tabla en la que cada fila representa una celda y donde cáda
columna representa el valor de esa variable para esa celda.
res <- as.data.frame(rb) %>%
dplyr::select("elevation", "rix", "power_dens_height.50", "JRC_1K_POP_2018")
plot(res)
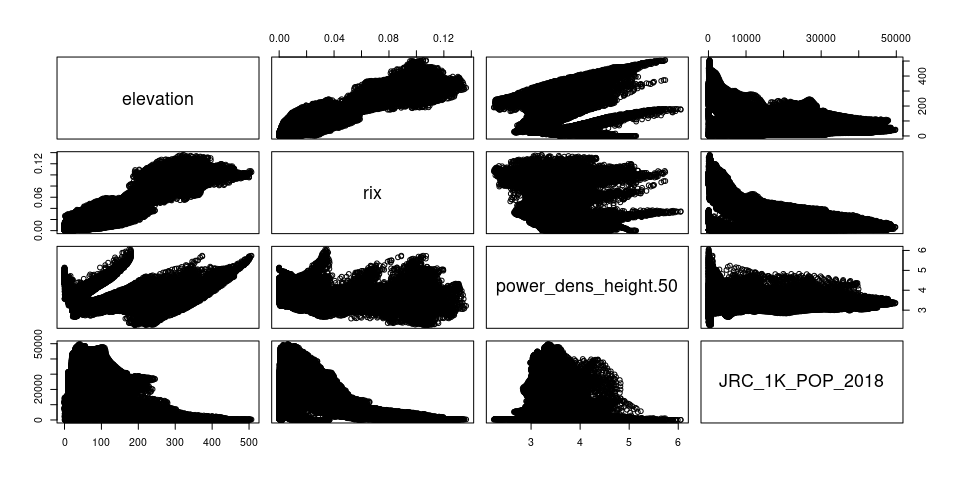
De esta forma tan sencilla, se podrían cruzar los datos encapsulados en
ese archivo .nc inicial descargado de la página del proyecto
*NEWA** con datos geográficos de cualquier otro tipo.
Te recomiendo que le des una vuelta y, si tienes alguna duda ya sabes donde encontrarnos.
-
Cita: Olsen, Bjarke Tobias; Davis, Neil; Hahmann, Andrea N.; Badger, Jake; Mann, Jakob; Vasiljevic, Nikola (2021): New European Wind Atlas: Microscale Atlas. Technical University of Denmark. Dataset. https://doi.org/10.11583/DTU.14672049.v1 ↩